K-Means en R
Importar datos:
datos <- read.csv("EAM_2019-v2.csv", sep = ";", dec = ",", header = T)
print(head(datos))
ï..gasto_personal inversion_AF ventas
1 32334949 92544622 321070109
2 5669798 69317638 86597330
3 11081213 50853587 26727389
4 47892609 42426113 388005721
5 56410101 35164028 390070138
6 20536798 27028346 490639882
dim(datos)
- 216
- 3
str(datos)
'data.frame': 216 obs. of 3 variables:
$ ï..gasto_personal: int 32334949 5669798 11081213 47892609 56410101 20536798 14001910 38533922 21397321 3478034 ...
$ inversion_AF : int 92544622 69317638 50853587 42426113 35164028 27028346 25150313 23019658 22007282 21924323 ...
$ ventas : int 321070109 86597330 26727389 388005721 390070138 490639882 379365700 488079500 233432780 9932602 ...
colnames(datos) <- c("Gasto_personal", "Inversión_AF", "Ventas")
Creamos una variable para almacenar solo las variables que queremos analizar.
df <- datos[, c("Gasto_personal", "Ventas")]
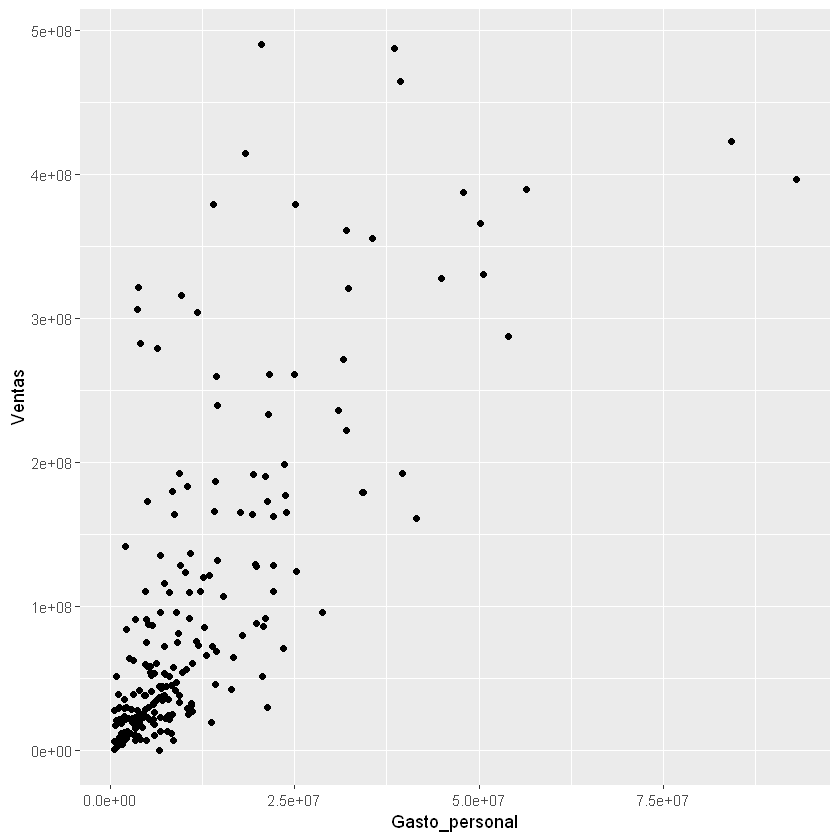
Dispersión de los datos:
library(ggplot2)
ggplot(data = df)+
geom_point(aes(x = Gasto_personal, y = Ventas))
Escalamiento de variables:
Para Normalizar usaremos la función scale().
df_scaled <- scale(df)
df_scaled <- data.frame(df_scaled) # Data Frame para ggplot2
print(head(df_scaled))
Gasto_personal Ventas
1 1.52915807 1.9822451
2 -0.47700945 -0.1241207
3 -0.06987862 -0.6619571
4 2.69964731 2.5835553
5 3.34046558 2.6021008
6 0.64151759 3.5055604
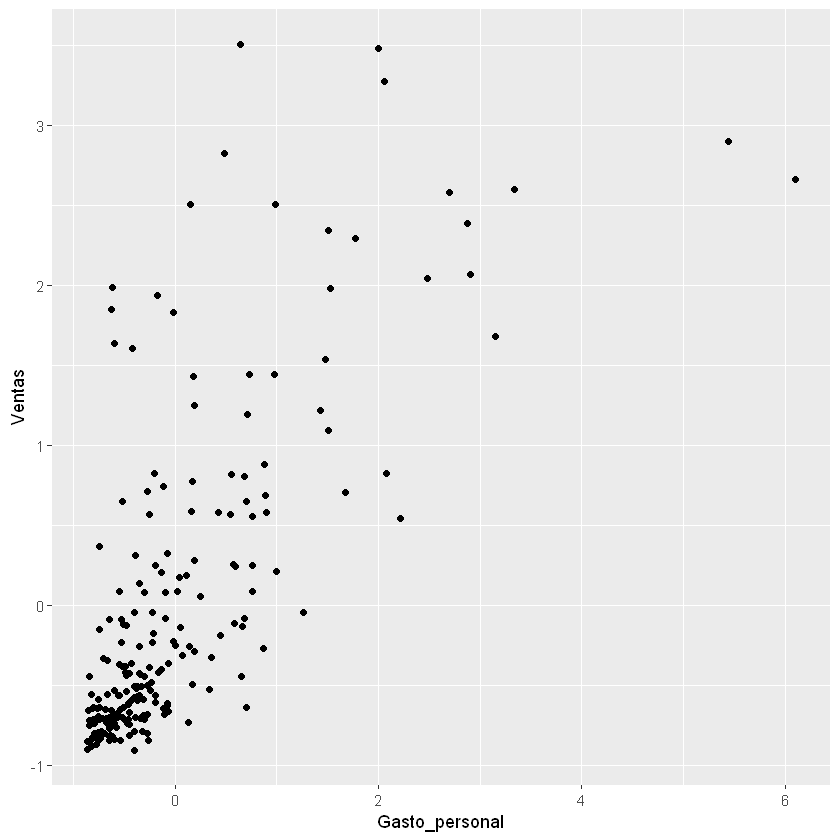
Dispersión de los datos escalados:
ggplot(data = df_scaled)+
geom_point(aes(x = Gasto_personal, y = Ventas))
Distancia Euclideana:
Con la función dist() podemos calcular diferentes distancias con el
argumento method =: "euclidean", "manhattan",
"minkowski", entre otros. Cuando se usa la distancia "minkowski"
se debe agregar el argumento p =.
dist_eucl <- dist(df_scaled, method = "euclidean")
Matriz de distancias:
De forma matricial se muestran las distancias entre todas las observaciones. La diagonal es cero porque es la distancia entre cada observación con ella misma.
Por ejemplo: la distancia Euclidiana entre la observación uno y dos es 2,9. Resta al cuadrado de la fila 1 con la fila 2 y el resultado se saca raíz cuadrada así:
print(sqrt((df_scaled[1,1]-df_scaled[2,1])^2+(df_scaled[1,2]-df_scaled[2,2])^2))
[1] 2.908863
Se sacará la matriz para las distancias entre las primeras 10 observaciones.
print(round(as.matrix(dist_eucl)[1:10, 1:10], 1))
1 2 3 4 5 6 7 8 9 10
1 0.0 2.9 3.1 1.3 1.9 1.8 1.5 1.6 1.1 3.5
2 2.9 0.0 0.7 4.2 4.7 3.8 2.7 4.4 1.8 0.7
3 3.1 0.7 0.0 4.3 4.7 4.2 3.2 4.6 2.0 0.6
4 1.3 4.2 4.3 0.0 0.6 2.3 2.6 1.1 2.4 4.8
5 1.9 4.7 4.7 0.6 0.0 2.8 3.2 1.6 3.0 5.2
6 1.8 3.8 4.2 2.3 2.8 0.0 1.1 1.4 2.3 4.5
7 1.5 2.7 3.2 2.6 3.2 1.1 0.0 2.1 1.4 3.4
8 1.6 4.4 4.6 1.1 1.6 1.4 2.1 0.0 2.6 5.0
9 1.1 1.8 2.0 2.4 3.0 2.3 1.4 2.6 0.0 2.4
10 3.5 0.7 0.6 4.8 5.2 4.5 3.4 5.0 2.4 0.0
K-Means:
La función más usada es kmeans() de la librería stats.
Instalar el siguiente paquete: install.packages("stats")
sintaxis: kmeans(x, centers, iter.max = 10, nstart = 1)
x: Data Frame con los datos, agregaremos las distancias calculadas
con dist().
centers: son los \(k\) clusters iniciales.
iter.max: número máximo de iteraciones. Por defecto es 10.
nstart: número de particiones iniciales aleatorias cuando
centers es un número. Probar con nstart > 1
set.seed(1) # valor semilla para obtener siempre los mismos resultados.
names(kmeans(dist_eucl, 10))
- 'cluster'
- 'centers'
- 'totss'
- 'withinss'
- 'tot.withinss'
- 'betweenss'
- 'size'
- 'iter'
- 'ifault'
cluster: un vector de enteros (desde 1:k) que indica el cluster al
que se asigna cada punto.
centers: una matriz con los centroides.
withinss: vector con los WCSS de cada cluster. El resultado es
después de definir la cantidad de clusters.
size: cantidad de puntos en cada cluster.
Número óptimo de clusters:
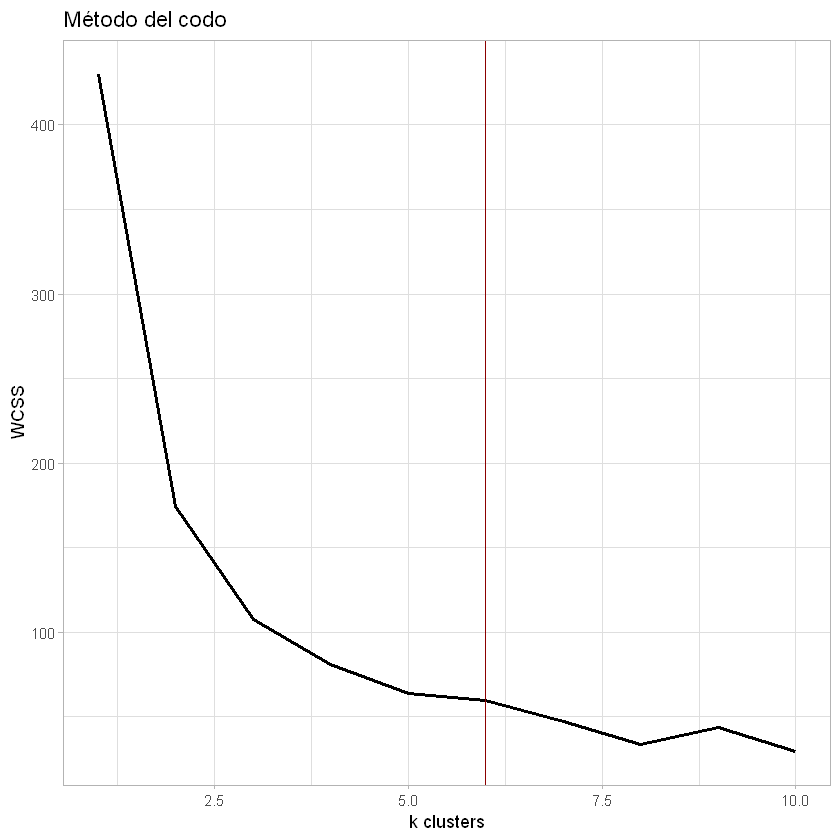
Método del codo:
Se calculará el valor de WCSS aplicando K-Means aumentando la cantidad de centroides.
wcss = vector()
for (i in 1:10){
wcss[i] <- sum(kmeans(df_scaled, i)$withinss)
}
ggplot()+geom_line(aes(x = c(1:10), y = wcss), size = 1)+
geom_vline(xintercept = 6, color = "darkred")+
labs(title = "Método del codo",
x = "k clusters",
y = "WCSS")+
theme_light()
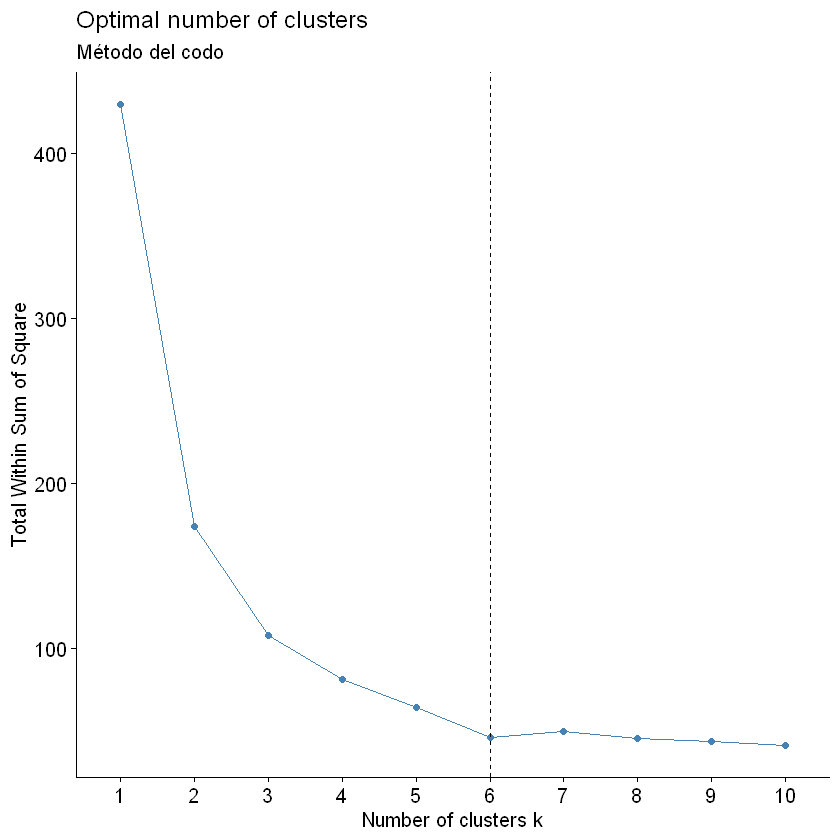
Este método también lo podemos hacer con la función fviz_nbclust()
del paquete factoextra.
install.packages("factoextra")
library(factoextra)
Warning message:
"package 'factoextra' was built under R version 4.1.3"
Welcome! Want to learn more? See two factoextra-related books at https://goo.gl/ve3WBa
fviz_nbclust(df_scaled, kmeans, method = "wss") +
geom_vline(xintercept = 6, linetype = 2)+
labs(subtitle = "Método del codo")
Método de la silueta:
Este método también lo hacemos con la función fviz_nbclust()
fviz_nbclust(df_scaled, kmeans, method = "silhouette")+
labs(subtitle = "Método de la silueta")

Método del gap estadístico:
fviz_nbclust(df_scaled, kmeans, method = "gap_stat")+
labs(subtitle = "Gap statistic method")

Aplicación del K-Means con k óptimo:
\(k = 3\):
Recomiendo llamar el modelo de cualquier forma diferente a kmeans
por este nombre se utiliza para algunas librerías como función y no como
una variable; por ejemplo, guardemos el resultado con el nombre de
k_means.
k_means <- kmeans(dist_eucl, 3, iter.max = 300, nstart = 10)
Cantidad de observaciones por cada cluster:
¿Cómo cambia este resultado para 3 clusters?
print(k_means$size)
[1] 15 46 155
Para visualizar los clusters en los datos originales usaremos la función
clusplot() del paquete cluster.
Instalar el paquete: install.packages("cluster")
library(cluster)
Warning message:
"package 'cluster' was built under R version 4.1.3"
clusplot(df,
k_means$cluster, # el cluster que se le asigna a cada punto.
lines = 0, # para eliminar líneas en el gráfico.
labels = 4, # 4 para que ponga una etiqueta al número del cluster.
color = TRUE, # diferente colo para cada sector.
plotchar = FALSE, # con FALSE se quitan los símbolos diferentes para cada cluster.
span = TRUE, # TRUE para que represente el cluster con una elipse no como círculo.
main = "Clustering de empresas",
xlab = "Activos Totales",
ylab = "Ingresos Operacionales"
)

Otra forma de visualizar los cluster con fviz_cluster()
fviz_cluster(k_means, data = df,
stand = FALSE, # FALSE para que no estandarice los datos, ya se había hecho esto.
show.clust.cent = TRUE, # TRUE para que muestre los centroides.
ellipse = TRUE, # Puede ser FALSE o TRUE
ggtheme = theme_minimal())
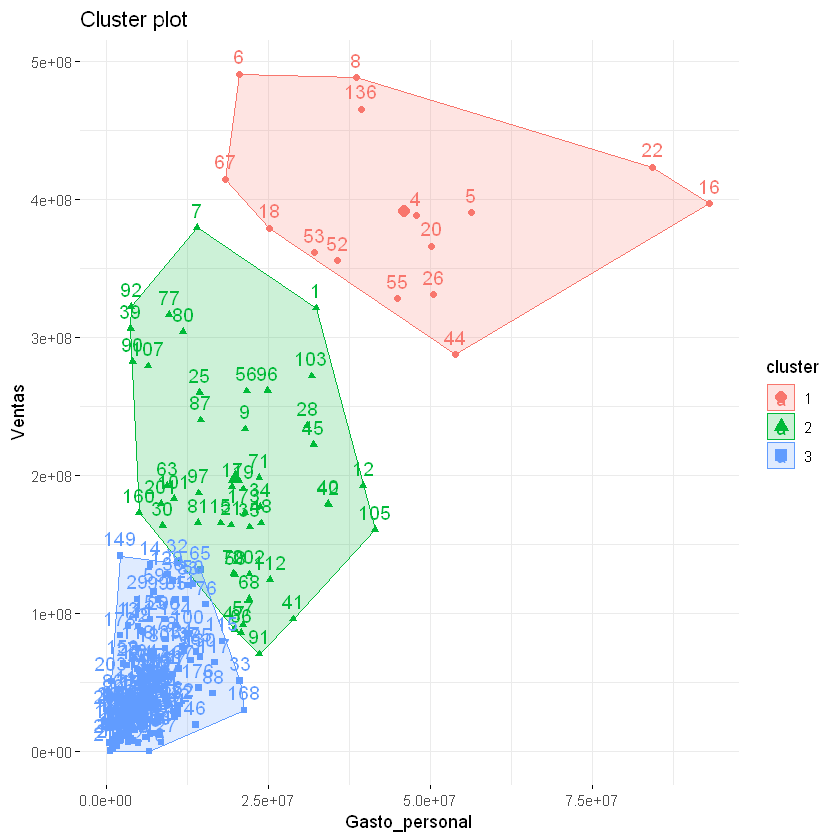
Es posible calcular la media de cada variable por cluster utilizando los datos originales:
print(aggregate(df, by = list(cluster = k_means$cluster), mean))
cluster Gasto_personal Ventas
1 1 46055433 390956062
2 2 19831448 197627597
3 3 6394092 43446519
\(k = 6\):
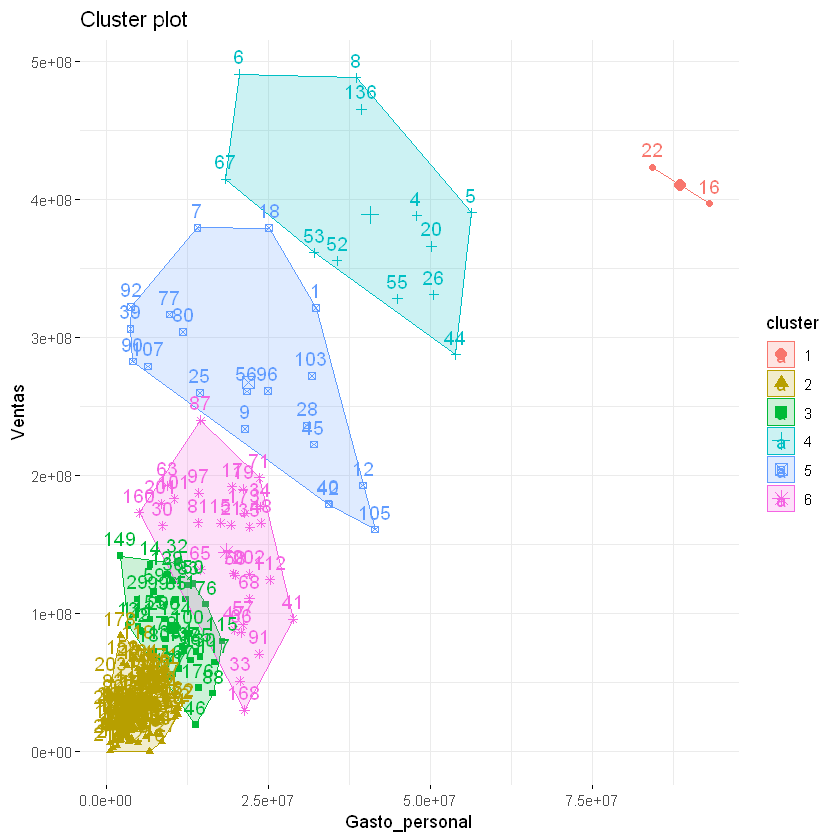
k_means <- kmeans(dist_eucl, 6, iter.max = 300, nstart = 10)
print(k_means$size)
[1] 2 115 37 12 20 30
fviz_cluster(k_means, data = df,
stand = FALSE, # FALSE para que no estandarice los datos, ya se había hecho esto.
show.clust.cent = TRUE, # TRUE para que muestre los centroides.
ellipse = TRUE, # Puede ser FALSE o TRUE
ggtheme = theme_minimal())
Realizar el K-Means con distancias Manhattan y Minkowski con p igual 0.1, 0.5, 3 y 4.
Realizar lo anterior, pero sin estandarizar las variables
¿Para este caso en específico será necesario la estandarización de variables?
Realizar el K-Means con las siguientes variables: Inversión en AF y Ventas