Escalamiento Multidimensional el R
Si se cuenta con multiples variables que describen las caracteristicas del problema pero se desea reducir la dimensionalidad, tal como pasaba con componentes principales o análisis factorial, pero esta vez tomando en cuenta la similitudes de los datos (como en clustering), puede hacer uso del escalamiento multidimensional. Se puede implementar tanto métrico como no métrico.
Métrico clásico:
Se busca conservar las distancias métricas originales, es el método recomendado para usarse con variables cuantitativas. También se conoce como el método de coordenadas principales.
En R, podemos utilizar las funciones cmdscale() de la librería
{stats} o también se puede hacer desde isoMDS() y
sammon()del paquete {MASS}
Leemos los datos, la BD utilizada es “EAM_2019_group.csv”
datos <- read.csv("EAM_2019_group.csv", sep = ",", dec = ",", header = T)
print(head(datos))
X datos.gasto_personal datos.costos_gastos_produccion datos.ventas
1 1011 1459203 177274 18565287
2 1020 814349 3566733 20658258
3 1030 7334998 5941761 115741258
4 1040 4959474 4536304 90612765
5 1051 9352991 6846304 192609248
6 1061 3720876 3799372 306175590
datos.inversion_AF
1 649040
2 1389081
3 5615593
4 1325824
5 4979745
6 9086434
Los datos deben contener etiquetas por lo que se le asigna a las filas los nombres de la primera columna
row.names(datos) <- datos$X
datos$X <- NULL
print(str(datos))
'data.frame': 60 obs. of 4 variables:
$ datos.gasto_personal : int 1459203 814349 7334998 4959474 9352991 3720876 47892609 11081213 21648490 10934460 ...
$ datos.costos_gastos_produccion: int 177274 3566733 5941761 4536304 6846304 3799372 44427434 1843527 12959271 16959038 ...
$ datos.ventas : int 18565287 20658258 115741258 90612765 192609248 306175590 388005721 26727389 260836023 136493373 ...
$ datos.inversion_AF : int 649040 1389081 5615593 1325824 4979745 9086434 42426113 50853587 5970883 11555678 ...
NULL
Calculamos las distancias Euclideas:
dist <- dist(datos, method = "euclidean")
Se ajusta el modelo de escalamiento métric, partiendo de las disimilitudes hasta llegar a un conjunto de coordenadas principales:
escalado <- cmdscale(dist)
Por defecto la función genera 2 grupos, existen autores que no recomiendan usar mas de 2, sin embargo, hay opiniones como Kruskal en 1964, que indica que máximo 9 dimensiones pueden ser usadas. la principal ventaja de este modelo su adpatabilidad a las necesidades del problema.
print(head(escalado))
[,1] [,2]
1011 -78710541 -2693081
1020 -76399117 -1226945
1030 18905169 -5253596
1040 -6616521 -7426404
1051 95360889 -13430884
1061 207837628 -25576966
Visualización de las nuevas coordenadas
library(ggrepel)
Warning message:
"package 'ggrepel' was built under R version 4.1.3"
Loading required package: ggplot2
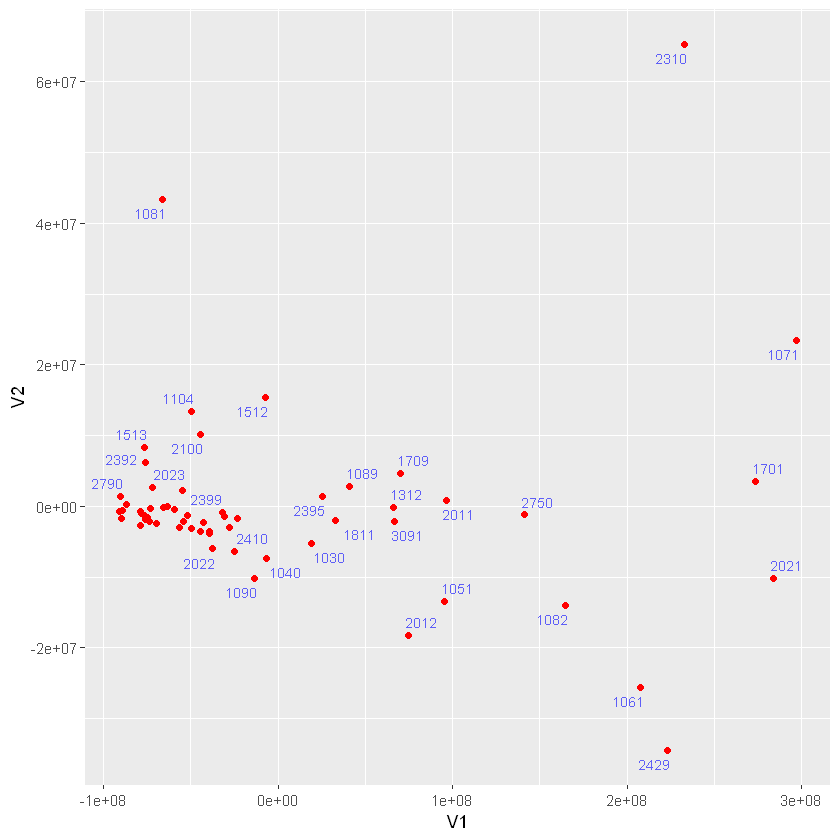
cmd_eam <- as.data.frame(escalado) #crea el dataframe, a partir de datos escalados
g1 <- ggplot(cmd_eam, aes( x = V1,
y = V2,
label = rownames(datos)))+
geom_point(color = "red")+
geom_text_repel(alpha = 0.6, size = 3, col = "blue") #grafica sin solapar los textos (omite los datos solapados)
print(g1)
Warning message:
"ggrepel: 32 unlabeled data points (too many overlaps). Consider increasing max.overlaps"
Escalamiento no métrico:
La función isoMDS() porporciona el algoritmo para realizar
escalamiento no métrico, sin embargo, si no le se proporciona la función
de rangos de los datos, realizará su solución por medio del método
clásico cmdscale(). Por tanto, el resultado será el mismo, ya que
k=2 por defecto.
library(MASS)
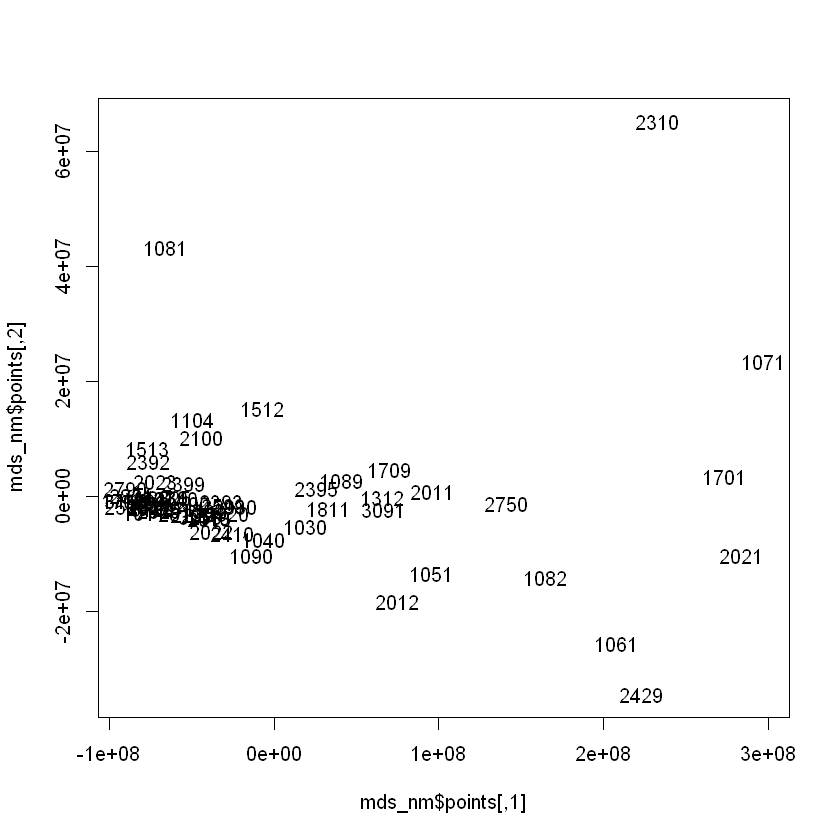
mds_nm <- isoMDS(dist) #ajusta el modelo clásico a la matriz de distancias.
plot(mds_nm$points, type = "n")
text(mds_nm$points, rownames(mds_nm$points))
initial value 1.881156
final value 1.881156
converged
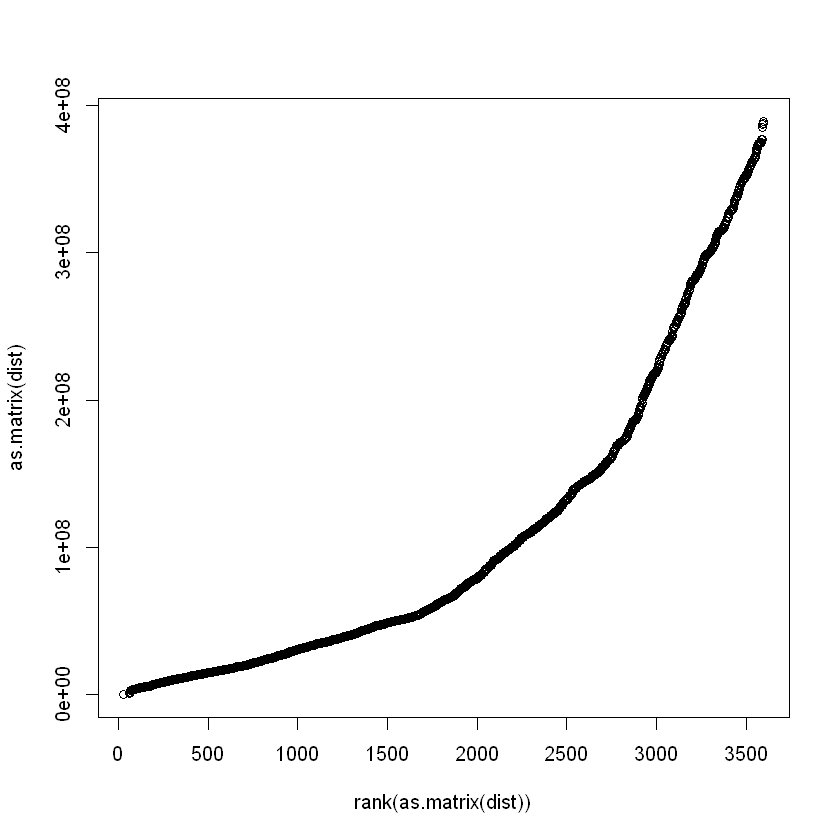
Para cambiar esta solución, debe realizarse el ranking u ordenamiento de
las variables. la función rank()de la base, proporciona esta
salida.
plot(rank(as.matrix(dist)), as.matrix(dist)) #la función Rank ordena las distancias como una sequiencia
empresa_rank <- matrix((rank(as.matrix(dist))), nrow = nrow(datos))# convierte en matriz
mds_nmrk2 <- isoMDS(empresa_rank) #aplica EMD a los datos ordenados
initial value 12.063241
final value 12.063215
converged
Visualizando el nuevo agrupamiento:
modelk2 <- as.data.frame(mds_nmrk2$points)#convierte la salida en DF para ser usada en ggplot2
g2 <- ggplot(modelk2, aes(x = V1,
y = V2,
label = rownames(datos)))+
geom_point(color = "red")+
geom_text_repel(alpha = 0.6, size = 3, col = "blue")
print(g2)
Warning message:
"ggrepel: 17 unlabeled data points (too many overlaps). Consider increasing max.overlaps"
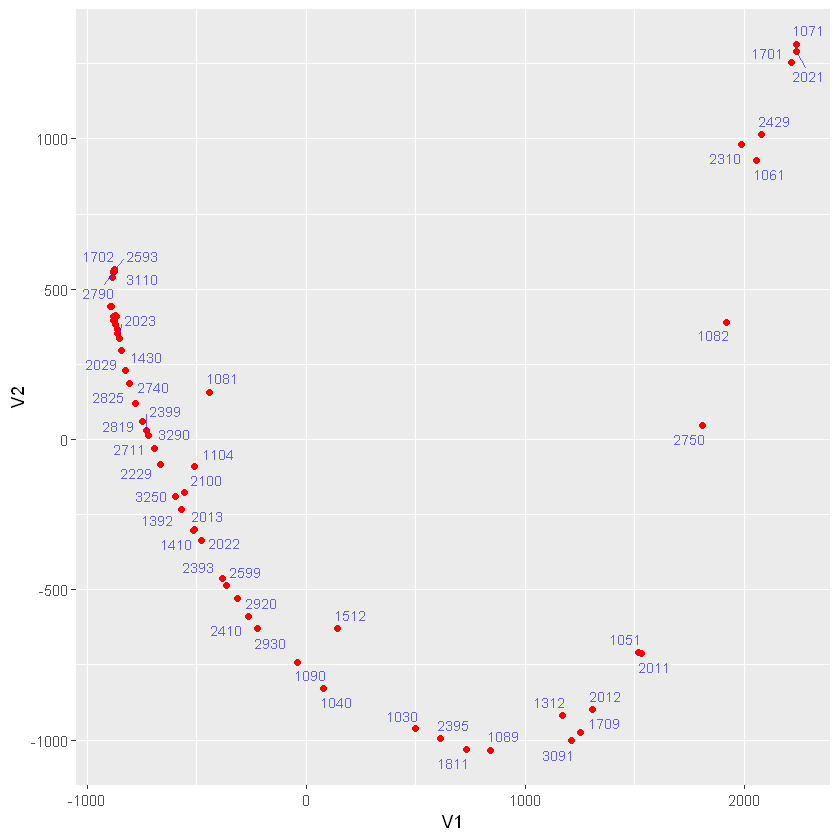
Diferencia entre métrico y no métrico
Se puede observar como el agrupamiento toma un orden dentro los dos rangos.
library(gridExtra)
grid.arrange(g1, g2, ncol = 2)
Warning message:
"package 'gridExtra' was built under R version 4.1.3"
Warning message:
"ggrepel: 35 unlabeled data points (too many overlaps). Consider increasing max.overlaps"
Warning message:
"ggrepel: 17 unlabeled data points (too many overlaps). Consider increasing max.overlaps"

Determinar el K óptimo:
Similar a los gráficos de sedimentación realizados en los otros modelos, se espera escoger aquel que modelo que lo minimice. Para ello se realiza el ajuste de diferentes K, donde se evalua el punto donde se logre estabilidad. Recuerde que según la teoría no es recomendable, realizar mas de 9 dimensiones.
#Ajuste para cada K en los datos ordenados
mds_nmrk1 <- isoMDS(empresa_rank,k=1)
mds_nmrk2 <- isoMDS(empresa_rank) #por defecto usa K=2
mds_nmrk3 <- isoMDS(empresa_rank, k=3)
mds_nmrk4 <- isoMDS(empresa_rank, k=4)
mds_nmrk5 <- isoMDS(empresa_rank, k=5)
mds_nmrk6 <- isoMDS(empresa_rank, k=6)
initial value 19.798271
final value 19.798202
converged
initial value 12.063241
final value 12.063215
converged
initial value 11.765570
final value 11.765554
converged
initial value 9.928388
final value 9.928374
converged
initial value 9.172647
final value 9.172632
converged
initial value 7.040586
final value 7.040566
converged
Gráfico de sedimentación del STRESS
stress = c(mds_nmrk1$stress, mds_nmrk2$stress, mds_nmrk3$stress, mds_nmrk4$stress, mds_nmrk5$stress, mds_nmrk6$stress)
dimensions = 1:6
plot(dimensions, stress, type = "b", xlab = "Number of Dimensions", ylab = "Stress")
print(stress)
[1] 19.798202 12.063215 11.765554 9.928374 9.172632 7.040566
